細胞内で生じる突然変異の脅威と日々戦うミスマッチ修復機構
1.はじめに
私たちの体を作るための設計図とも言える遺伝情報DNA(デオキシリボ核酸)は、常に突然変異(遺伝情報が変化すること)の脅威にさらされており、その要因は大きく分けて2つあります。一つは、細胞増殖においてDNAが合成(複製)される時など細胞の活動に伴って生じる内的要因、もう一つは、放射線、紫外線そして化学物質ばく露などによる外的要因です。本コラムでは、前者、特に "DNA複製"がもたらし得る脅威や、その際に生じた変異を検出し、元の遺伝情報に直す仕組み "ミスマッチ修復"を中心に解説したいと思います。ミスマッチ修復経路が正常に機能しない場合、がんを伴うリンチ症候群を発症することや、ある種の抗がん剤の効き方(奏功率)に変化が生じる点についても触れ、最後に、これらの知見が、筆者が現在行っている産業化学物質の毒性メカニズム解析にどう生かして行けるのかについて述べたいと思います。
2.DNA複製は、チャレンジングなプロセス
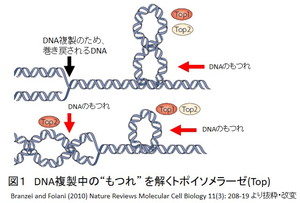
両親から半分ずつ受け継がれた遺伝情報の本体は、デオキシリボースとリン酸に、4種類の塩基:アデニン(A)、グアニン(G)、シトシン(C)、チミン(T)が幾重にも連なって出来たDNAですが、AはTと、CはGと相補的な対を作り、2重らせんを作っています(発見者らにちなんでワトソン-クリック対と呼ばれます)。体を構成する細胞は活動上、必要に応じて増殖しますが、その過程でDNAは倍加(複製)します。その作業は元のDNA情報を読み取りながら行われるため、2重らせんを巻き戻す必要があります。DNAヘリカーゼ(MCM複合体)がその役割を担いますが、その際に、らせんを解くことでDNAにもつれ(ねじれ)が生じてしまいます(図1と文献1)。それらは、DNAトポイソメラーゼ(Top)により切断され、すぐに再結合されることで解消されながら(図1)、DNAポリメラーゼはDNAの配列を一定方向に読み取り、A、T、G、Cを含む原材料を挿入しDNAを複製します。
複製ポリメラーゼは非常に正確ですが、109個の塩基対と非常に長いヒトゲノムDNAを合成する途中、当然のことながら誤った塩基を挿入してしまう場合があります。しかし興味深いことに、DNAポリメラーゼは"校正機構"を備えているので、誤りは削られ、その部分が再合成されることで、エラー率は1/107にまで下がると言われています。これをさらに強化し、ゲノム安定性を維持するために、細胞はミスマッチ修復というバックアップ機構を持っています。筆者には、前所属機関においてミスマッチ修復の研究に従事していた経緯もあるので、次にその詳細について紹介します。
3.DNA複製中に残ったエラーを直す、ミスマッチ修復経路
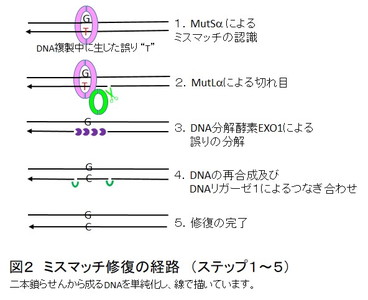
前節で述べたDNAポリメラーゼの校正機構をすり抜けた塩基対間の誤り(非ワトソン-クリック対)に加えて、AAAやTTTの様な繰り返し配列においてDNAポリメラーゼのスリップにより生じる塩基付加や塩基欠損は、ミスマッチ修復経路により認識・修復され、誤りはさらに 1/1000程に低下します(文献2)。ミスマッチ修復を含むDNA修復経路の詳細を解明したPaul Modrichら3博士には、2015年にノーベル化学賞が贈られています。
ミスマッチ修復経路では、まずMutSα(MSH2とMSH6の二つの異なるタンパク質からなる二量体)が誤りを含むDNAと相互作用し、両手でDNAを取り囲む様にして誤りを認識します(図2)。さらにMutLα(MLH1とPMS2の二つの異なるタンパク質からなる二量体)と結合し複合体を形成します。MutLαを構成するPMS2が誤った塩基の近傍のDNAに"切れ目"を入れ、誤りを含むDNA鎖を取り除くための準備を始めます。この発見は、ヒトのミスマッチ修復メカニズム解明における一つの突破口と言え、上記のModrich博士の研究室から報告されています(文献3)。この切れ目を利用してリクルートされたDNA分解酵素EXO1が誤りを含むDNAを分解し、再度DNAが合成されます。最終的に、DNAリガーゼ1により新規合成部分のDNAがつなぎ合わされ、修復は完了します。現在知られているDNA分解酵素はEXO1のみですが、それ以外の酵素が機能する可能性も示唆されており(文献4)、今後の詳細解明が期待されます。
4.ミスマッチ修復経路の遺伝子変異により生じるリンチ症候群
MutSαやMutLαは、DNAの情報を基に作られる各2種類のタンパク質からなりますが、これらをコードしている遺伝子に変異が生じる場合があります。もしこれらの遺伝子における変異を遺伝的に受け継いだ場合、若年性がんを発症するリンチ症候群を発症することが知られています(文献2)。臨床においては、MutSαやMutLαを構成するMSH2やMLH1の発現低下に加え、ミスマッチ修復経路の機能欠損で顕著となるAAAやTTTの様な“繰り返し配列の延長” (マイクロサテライト不安定性と呼ばれます)により診断が行われます。上記以外にも、ミスマッチ修復遺伝子の働きを制御するプロモーター部位のメチル化等による機能低下が起こった場合でも(家族性でない)散発的がんが引き起こされることから、この修復経路の重要性が分かります。
5.MNUなどの抗がん剤の奏功にはミスマッチ修復経路が必要
がん細胞は正常細胞より早く増殖する場合が多いことから、DNA複製は、抗がん剤の重要な標的となります。そのため、複製中のDNAのもつれを解くトポイソメラーゼ(Top)阻害剤は、重要な抗がん剤の一つです(文献1)。以下に紹介するアルキル化剤の一種ニトロソウレア(MNU)は、ミスマッチ修復機能により抗がん剤としての効果が得られますが(文献2)、その詳細は以下の通りです。
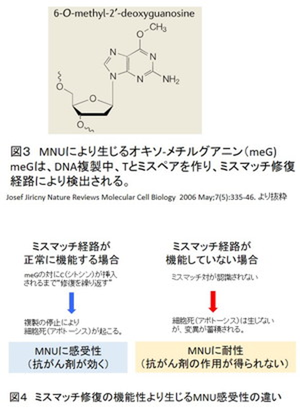
複製中にDNAに取り込まれたMNUは、細胞毒性を生じさせるオキソ-メチルグアニン(meG)を誘導します (図3)。meGは、DNA複製の際にCとのペアと同等にTとミスペアを作るため、meG-Tはミスマッチ修復経路により認識されます。重要なのは、この修復経路は、複製元のDNA鎖ではなく新合成されたDNA鎖を修復する特徴を持っていることです。そのため親鎖のmeGではなく、新生鎖にあるTの除去が試みられます。しかし、これはmeGを取り除くという根本的な修復ではないため、Tの除去→DNA再合成→DNAがつながれる という一連の修復過程が、シトシン(C)が挿入されるまで"何度も"行われます(文献2と図4)。そのうち複製は停止し、細胞死(アポトーシス)が誘導され、抗がん剤としての効き目が現れます。一方、ミスマッチ修復機能が欠損した細胞では、meG-Tのミスペアは認識されず、修復経路は機能しないため、ミスマッチ修復が正常である野生型のヒト培養細胞と比べると、100倍ほど耐性となり、抗がん剤の効果が現れない状態となります(図4)。さらに、修復されないことで変異が誘導されてしまいます。そのため、MNUをがん治療で使用するときは、ミスマッチ修復遺伝子が機能することが奏功率の鍵を握ります。また細胞は、MGMT(O6-methyl guanine DNA methyl transferase)と呼ばれる、直接meGを取り除く酵素を有しており、その発現が非常に高い場合などは、MGMT阻害剤をMNUと同時に処置することが必要となります。
6.最後に
現在、産業化学物質は約7万種類あると言われていますが、労働者の安全に配慮した上で利用するには、化学物質の毒性評価や毒性メカニズムに関する情報の蓄積が不可欠です。今回紹介したDNA修復経路の機能性と薬剤感受性の関連についての知見は、産業化学物質の毒性メカニズム解析に利用できる可能性があります。例えば、DNA損傷性を有する修復経路不明の化学物質について、ミスマッチ修復遺伝子が正常または欠損した培養細胞を用い、その化学物質への感受性を比較します。そうすることで、その化学物質とミスマッチ修復経路との関連がわかります。MNUの例のように耐性を示した場合は、変異原性がある可能性も考えられます。現在では、ミスマッチ修復経路以外にも、多くのDNA修復経路の機能性と薬剤感受性との関連が分かっており(文献5、6)、主要なDNA修復経路の遺伝子欠損細胞を用いることで、化学物質の毒性機序が明らかにできる可能性があります。現在筆者は、この手法を取り入れた産業化学物質の毒性メカニズム解析を行っており、今後、その結果についての情報を発信して行きたいと考えています。
引用文献
- Dana Branzei and Marco Foiani (2010) Maintaining genome stability at the replication fork, Nat Rev Mol Cell Biol 11(3):208-19.
- Josef Jiricny (2006) The multifaceted mismatch-repair system, Nat Rev Mol Cell Biol 7(5):335-46.
- Farid A Kadyrov et al., (2006) Endonucleolytic function of MutLalpha in human mismatch repair, Cell 28;126(2):297-308.
- Amar Desai and Stanton Gerson (2014) Exo1 independent DNA mismatch repair involves multiple compensatory nucleases, DNA Repair (Amst).21: 55-64.
- Junko Murai et al., (2012) Trapping of PARP1 and PARP2 by Clinical PARP Inhibitors, Cancer Res. 1;72(21).
- Yuko Maeda et al., (2014) Differential and common DNA repair pathways for topoisomerase I- and II-targeted drugs in a genetic DT40 repair cell screen panel, Mol Cancer Ther.13(1):214-20.